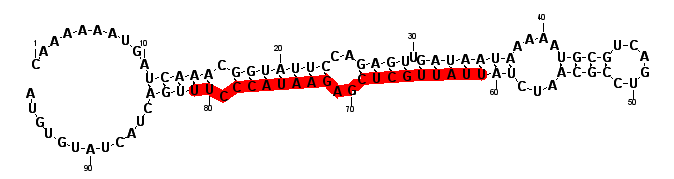
| seed | ------------------------------------------------------------------------------UAUUGCU--------------------------------------------- | 25 | miR-234 |
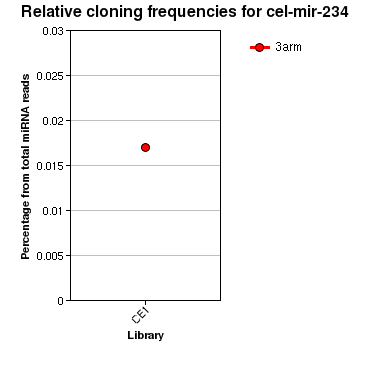
| | | | CE1 |
| cel-miR-234 | -----------------------------------------------------------------------------TTATTGCTCGAGAATACCCTT-------------------------------- | 21 | 23 |
| | -----------------------------------------------------------------------------TTATTGCTCGAGAATACCCT--------------------------------- | 20 | 2 |
| celegans | ----------------CAAAAAATGATCAAACGGTATTCCAGAGTTGATAATAAAAATG-CGTCAGTCCGCAATCTATTATTGCTCGAGAATACCCTTTGAC------TACTATGTGTA----------- | |
| cbriggsae | -----------------------------AACGGTATTCCTGAGTGAATAATAAAAATG-------AGTCCATTCTATTATTGCTCGAGAATACCCTT-------------------------------- | |
| cjaponica | -----------AAAAACACACCACGTTCGATAGGTGTTCCAGAGTTGATAATAATCGTGCCAT--GTGCACATT-TATTATTGCTTGAGAATACCCCTCGGCAGATTGTGTTCTGTTTTT---------- | |
| cbrenneri | ------GAATTCATGCAACAATCAGCTCAAACGGTATTCTTTGGTTAATAATAAGGATGGCATCACGACTCATTATATTATTGCTCAAGAATACCCTTTGACAAATGTCGCGTGACATC----------- | |
| cremanei | GATGAATGGTAAGTGGTGAACCAAGCTCAAACGGTATTCGACGGTGAATAATAAAAATGACATTGCGACTCATTCTATTATTGCTCGAGAATACCCTTTGACACGGTTCACCTTTTTTTTGGTATTTGTC | |
| | * *** *** ** ******* ** ** * ********** ********* * | |
| celegans | ..........(((((.(((((((..((((.(((((((....(( ((......))))...)))))))))))..))))))).))))). ........... | 1.000 -25.80 |
| cbriggsae | ...(((((((.(((((.(((((((..(((( ....)))).)))))))))))).)))))))... | 1.000 -19.60 |
| cjaponica | (((((((...((((.((((..(((((((..(((.(((((((((..((((... ..))))..) )))))))))))..)))))))..)))).....))))...))))))) | 0.980 -32.00 |
| cbrenneri | ....((((((.(((......(((((.(((((((((.((.((((((((..(((((.........))))).)))))))))).))))))))).)))))....))).)))))).... | 0.999 -32.00 |
| cremanei | ((..((((.((((.((((((((....(((((.(((((((..((((.(((((((..(((((.........))))).))))))))).)).))))))).)))))...)))))))).....)))).))))..)) | 1.000 -40.40 |