| seed | ---------------------------------------------------------------------------------CACAGGA---------------------------------------------------- | 2 | miR-249 |
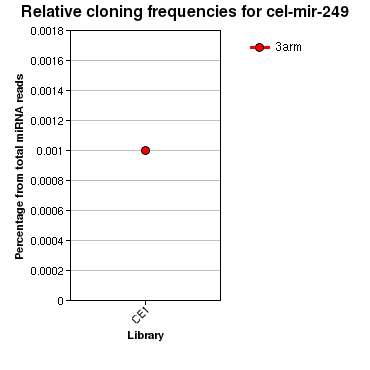
| | | | CE1 |
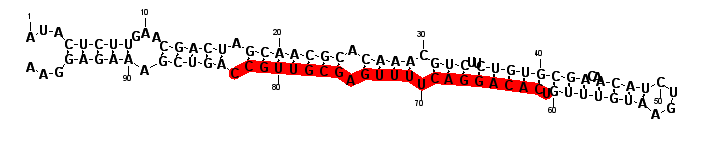
| cel-miR-249 | --------------------------------------------------------------------------------TCACAGGACTTTTGAGCGTTGCC------------------------------------- | 23 | 2 |
| celegans | -----------------ATACTCT-TGAACGACTAGCAACGCACAAACGTCTTCTGTGCGACAA--CATCTGAATGTTTGTCACAGGACTTTTGAGCGTTGCCAGTC---GAAAGAGGAA-------------------- | |
| cbriggsae | GCTATTTGTTATTTTGTGCGCCCTCAATGCTACTAGCAACGCTCAAAAAT-CACTGTACGACAAATGACT--ATATTTTGTCACAGGATTTTTGAGTGTTGCTAGTC---GGAGAAGGGCCGTCTTGAAATAATTCAGGT | |
| cjaponica | -----TTTTAAATTGTTTTCCTCTCCCTGT--CTAGCGGCGC-CAAAAGATTACTGTGAGGCAAAGAAATGGTGAGCTTGTCACAGGACTTTTGAGCGTTGCTAGTTATAGAGGATGGAATTGTAAGTTTTTGA------ | |
| cbrenneri | -CTAACTTTTATTTTTTTGTCTCTCTGAGCTACTAGCAACGC-TAGAAGTCTACTGTTAGACAGAGTACA-GAAATTTTGTCACAGGACTTTTGAGCGTTGCTGGTTAATGAGAGCCTATT--TTAGTAATTAGTAAG-- | |
| cremanei | TCTCATTACT-TCTATTTTGTTTTCTACGTGACTAGCAACGCACAAAAGT-TTCTGTGCGACAA---ATATGAAATTTTGTCACAGGACTTTTGAGCGTTGCTAGTT---GAAAGAAGATTCACTATGATCAGATCAGG- | |
| | * ***** *** * * **** * ** * *********** ******* ***** ** * | |
| celegans | ...(((( (...(((((.(((((((.((((.(((..((((((((..( (((....))))))).)))))))).)))).))))))).)))) ).)))))... | 1.000 -40.60 |
| cbriggsae | (((....(((((((((.(((((((.....((((((((((((((((((((( (.((((..((((((..... ....)))))))))))))))))))))))))))))). ))...))))).))..)))))))))...))) | 0.968 -54.70 |
| cjaponica | ....(((((...((((..((((((( (((((((((( ((((((....(((((((((.............)))..))))))..)))))).))))))))))..)))).))).)))).....))))).... | 0.993 -42.12 |
| cbrenneri | ...(((.(((((.......(((((......((((((((((( ((((((((..((((..((((((((... ...))))))))))))))))))).))))))))))))....)))))...... ..)))))..)))... | 0.987 -45.96 |
| cremanei | .((.(((... ((......(((((((...(((((((((((((.((((((. ((((((..((((( (........)))))))))))))))))).)))))))))))) )..))))))).......))...))).)). | 0.826 -45.32 |